2023年1月20日九游会老哥俱乐部、省部共建生物催化与酶工程国家重点实验室吴姗团队与华中农业大学作物遗传改良国家重点实验室、洪山实验室蛋白质科学殷平教授研究团队合作,在国际重要学术期刊(Cell Discovery)杂志在线发表题为“Structural insights into DNA N6-adenine methylation by the MTA1 complex”的研究论文。湖北大学生科院吴姗和华中农业大学殷平教授为该论文的共同通讯作者。湖北大学冷冻电镜平台为该研究的开展提供了支持。此项研究综合利用了人工智能,生物化学和结构生物学的技术手段对MTA1复合体介导的DNA 6mA的分子机制进行了研究。
DNA和RNA的甲基化在调节生物体的各种生物过程中起着关键作用。除了DNA C5-胞嘧啶甲基化(5mC)和RNA N6-腺苷甲基化(m6A),在原核生物和真核生物中发现的另一种普遍的甲基化类型是N6-甲基脱氧腺嘌呤(6mA)。纤毛状嗜热四膜虫中鉴定的MTA1复合物是第一个经实验验证的真核生物6mA甲基转移酶(MTases)复合物,由MTA1、MTA9、p1和p2四个亚基组成(图1)。然而MTA1四元复合物的整体结构和工作机制尚不清楚,对整个MTA1复合物的结构研究将有助于阐明这四个亚基的工作模式,更好地理解真核生物的6mA MTase。
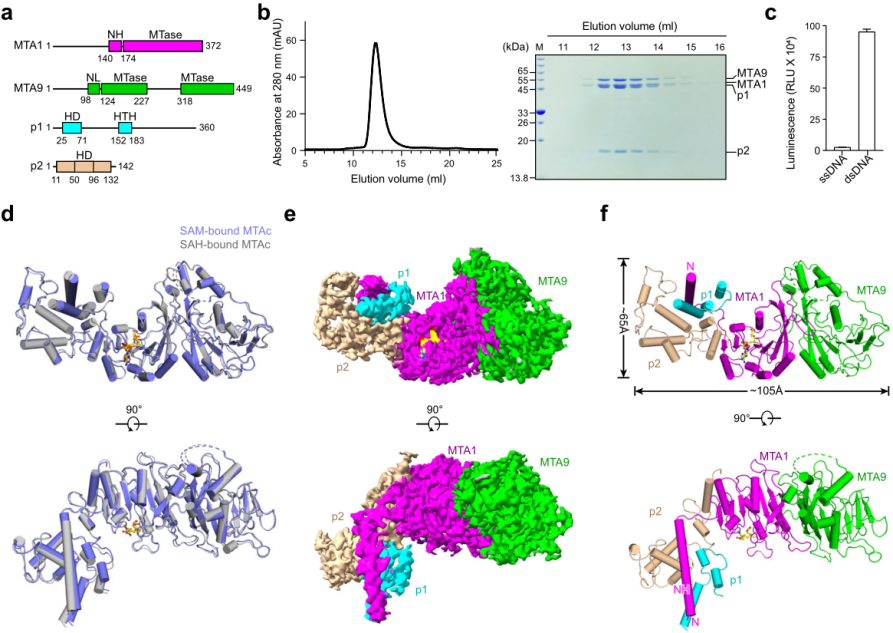
图1 辅因子底物结合MTA1复合物的整体结构
研究团队解析了S-腺苷蛋氨酸(SAM)结合态(2.6 Å)和S-腺苷同型半胱氨酸(SAH)结合态(2.8 Å)的MTA1四元复合物的冷冻电镜结构。在这两种结构中,MTA1, MTA9, p1, p2组成了一个化学计量比为1:1:1:1的异四聚体。结合结构分析和生化数据表征发现MTA1是容纳SAM/SAH的催化核心,MTA9和p1可促进 MTA1复合物与DNA结合,p2稳定 MTA1并帮助它结合甲基供体SAM。此外,MTA1复合物与哺乳动物m6A转移酶核心复合物METTL3–METTL14复合体的比对分析显示复合物中MTases 起源于相同的祖先“class-β MTase”,并且可能经历了进化分歧。这些复合物的MTases结构域相对保守,而它们的目标识别结构域TRD(可能是额外的调节亚基)则显示多样化,这些多样化的TRD可能决定复合物与其底物核酸的结合特异性。这些结果共同为MTA1复合物甲基化的分子机制和N6-腺嘌呤甲基化的MTase的潜在多样化提供了见解。
据悉,吴姗团队长期从事生物大分子的精细结构解析与相关分子机制研究,在Nature、Science、Nature Microbiology、Nature Structural & Molecular Biology、Nature Communications、Cell Discovery等国际知名期刊上发表多篇高水平文章。该研究得到科技部、国家自然科学基金委员会等的资助。
原文链接:https://www.nature.com/articles/s41421-022-00516-w




