近日,湖北大学九游会老哥俱乐部、省部共建生物催化与酶工程国家重点实验室李爱涛教授团队和郭瑞庭教授团队合作在催化领域Top期刊《ACS Catalysis》上发表题为“Rationally controlling selective steroid hydroxylation via scaffold sampling of a P450 family”(通过支架取样P450家族合理控制选择性类固醇羟基化)的研究性论文,并被选为封面文章(图1)。该研究挖掘得到一种新型细胞色素单加氧酶CYP109B4,首次解析了CYP109B4及其复合物的晶体结构,并基于此对其进行蛋白质理性设计,成功揭示了P450 CYP109B家族催化甾体选择性羟基化的分子调控基础,实现了其催化甾体底物C15β和C16β位选择性互换。博士生张小栋,沈盼盼和赵晶副教授为本文并列第一作者,郭瑞庭教授和李爱涛教授为通讯作者。

图1 杂志封面
对复杂甾体化合物进行羟基化,特别是在预定的惰性位点对甾体进行区域和立体选择性的羟基化,是合成有机化学中一个非常具有挑战性的反应,并且反应过程通常涉及高能耗、高污染、反应步骤繁琐等一系列问题。利用细胞色素P450在指定位置高选择性地对甾体进行羟基化修饰,因其高效率和原子经济性而受到越来越多的关注。目前已有许多真核生物P450s被报道能够催化甾体的羟基化。然而,真核生物P450s通常为膜结合蛋白,异源表达困难,催化活性低,因此严重阻碍了它们的工业应用。相比之下,细菌的P450s是可溶性酶,可以很容易地大量表达,并具有更高的生物转化活性。尽管如此,目前已报道的能够高效催化甾体羟基化的细菌P450s数量仍然有限。此外,这些细菌P450s羟基化甾体的选择性和活性分子调控机制还没有得到充分研究,因此,亟待发掘更多优良的细菌P450s,并开发高效定向改造技术,从而创制出高效的甾体羟基化酶,为甾体药物的高效生物合成提供绿色的生物催化过程。

图2. CYP109B4催化甾体羟基化的理性改造
了发掘新型的P450甾体羟化酶,该研究利用基因组挖掘技术,从索诺拉沙漠芽孢杆菌(Bacillus sonorensis)中分离得到了细胞色素P450单加氧酶CYP109B4,该酶对甾体底物睾酮具有良好的16β羟基化选择性和活性。利用X射线衍射技术对CYP109B4及其底物复合物的晶体结构进行解析,揭示了其16β选择性羟基化的分子基础,并鉴定了活性口袋中调控选择性和活性的关键残基I291,V292和S387。随后,基于CYP109B4与其家族成员CYP109B1结构与功能对比的差异,选定了潜在的关键氨基酸骨架,对其进行了理性设计。通过将突变景观设计(ML), 聚焦理性迭代位点特异性突变(FRISM)和迭代饱和突变(ISM)的突变策略相结合,成功将CYP109B4催化甾体羟基化的选择性从16β向15β转换(图2),实现了一系列甾体底物C16β和C15β的定向羟基化。此外,通过计算机模拟对CYP109B4的突变体进行分子对接和分子动力学模拟(MD),详细揭示了CYP109B4的突变体在分子水平上的高区域/立体选择性的分子催化机制。最后,通过对CYP109B家族的其它成员进行同源建模,并与CYP109B4的晶体结构进行叠合比对,选定结构上对应的关键氨基酸骨架进行定点突变,成功改变了它们的催化选择性,进一步证明了“热点”氨基酸介导的选择性调控(16β和15β之间)在CYP109B家族的普适性(图 3)。
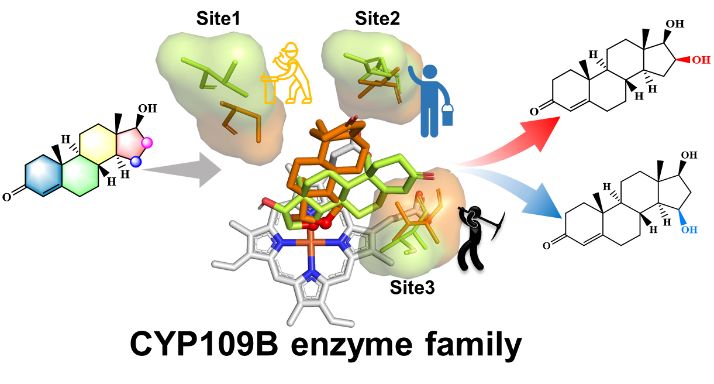
图3. 细胞色素P450酶蛋白骨架的理性设计调控其催化甾体的选择性羟基化
据悉,上述研究得到了科技部国家重点研发计划(2019YFA0905000 & 2019YFA0906400)、国家自然科学基金面上项目(21977026),湖北省杰出青年学者项目(2020CFA072)、湖北省学科引进人才创新基地项目(2019BJH021),以及省部共建生物催化与酶工程国家重点实验室自主研发基金等项目的资助。
文章链接:https://pubs.acs.org/doi/full/10.1021/acscatal.2c04906




